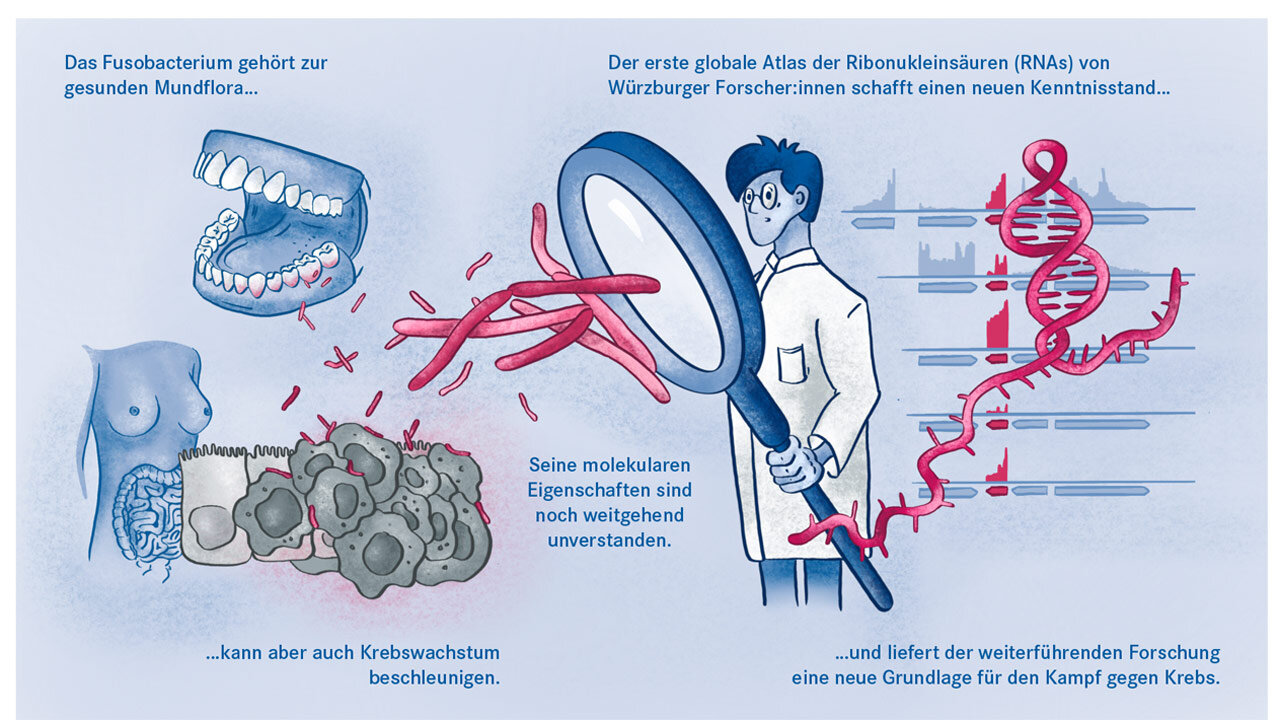
Der Mundhöhlenkeim Fusobacterium nucleatum ist dafür bekannt, das Wachstum menschlicher Karzinome, etwa im Darm oder in der Brust, zu beschleunigen. Das Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI), ein Standort des Helmholtz-Zentrums für Infektionsforschung (HZI), und die Julius-Maximilians-Universität (JMU) in Würzburg haben jetzt in einer gemeinsamen Studie die RNA-Moleküle von fünf klinisch relevanten Stämmen dieses anpassungsfähigen Erregers kartiert. Die gewonnenen Erkenntnisse könnten dazu beitragen, neue Therapien bei verschiedenen Krebserkrankungen zu entwickeln. Die Forschungsergebnisse wurden in der Fachzeitschrift Nature Microbiology veröffentlicht.
Der „fiese Fuso“
Das Fusobacterium nucleatum kommt in der menschlichen Mundhöhle vor und ist ein wichtiger Bestandteil der gesunden Mundflora. Es kann jedoch auch zur Entstehung von Parodontitis beitragen. Und noch wichtiger: Der Keim wird zunehmend auch innerhalb des menschlichen Körpers, nämlich auf Darmkrebszellen und Mammakarzinomen nachgewiesen. Dort scheint er das Tumorwachstum zu befördern und die Behandlung zu erschweren. Die bisherige Grundlagenforschung hat erste Erkenntnisse über die molekulare Reaktion der Wirtszellen auf die Bakterien erbracht. Weitgehend unverstanden sind bislang jedoch die molekularen Eigenschaften des Erregers selbst, seine Aktivität im Krebsgewebe und wie es ihm gelingt, sich nicht nur im Mund, sondern an ganz unterschiedlichen Stellen im menschlichen Körper anzusiedeln.
Wissenschaftlerinnen und Wissenschaftler vom HIRI in Würzburg sowie vom Institut für Molekulare Infektionsbiologie (IMIB) der JMU sind bei diesen Fragen jetzt einen entscheidenden Schritt vorangekommen. Sie haben für fünf klinisch relevante Stämme des Keims einen globalen Atlas der RNAs, also der Ribonukleinsäuren (von engl. ribonucleic acid) erstellt. Damit können sie hunderte zuvor unbekannte Ereignisse in der Genregulation der Mikroorganismen nachweisen und zeigen, wie sich diese im Laufe des Bakterienwachstums verändern. Außerdem haben die Forschenden erstmals einen Überexpressions-Vektor als genetisches Werkzeug im Fusobacterium eingesetzt, um die Funktionen seines Erbguts zu untersuchen.
RNA-basierter Ansatz eröffnet neue Möglichkeiten
„Unser RNA-basierter Ansatz hilft, einen klinisch relevanten Mikroorganismus auf molekularbiologischer Ebene zu analysieren und besser zu verstehen“, sagt HIRI-Direktor Jörg Vogel. Der Professor und Direktor des IMIB ist Initiator der aktuellen Studie und zuversichtlich, dass die vorliegenden Erkenntnisse die weiterführende translationale, also auf die medizinische Anwendung zielende Forschung befördern werden.
Die durchgeführte RNA-Kartierung umfasst Fusobacterium nucleatum mit seinen Unterarten nucleatum, animalis, polymorphum und vincentii sowie Fusobacterium periodonticum. Die Wissenschaftlerinnen und Wissenschaftler haben die primären Transkriptome – die Gesamtheit der RNA-Moleküle – dieser Stämme erfasst. Um deren genetische Funktionen zu untersuchen, setzte das Team einen Überexpressions-Vektor ein. Dieser kann ein bestimmtes Gen dauerhaft „anschalten“, um dessen Wirkweise genau zu analysieren. Durch den Einsatz des Überexpressions-Vektors wurden kleine regulatorische RNAs, sogenannte sRNAs (von engl. small RNAs) entdeckt, die eine regulatorische Funktion in den Mikroorganismen erfüllen. Eine durchaus interessante Beobachtung, wie Falk Ponath, Erstautor der Studie, meint: „Fusobacterium nucleatum hat sich in seiner evolutionsbiologischen Entwicklung recht früh von anderen Bakterien entfernt. Deswegen sind wir nicht von vornherein davon ausgegangen, in den untersuchten Stämmen sRNA zu entdecken, die ähnlich agiert wie bereits bekannte in anderen Bakterien.“ Und nicht nur das: Das Team konnte ebenso nachweisen, dass diese kleine RNA ein Protein der äußeren Zellmembran reguliert.
Die Studienergebnisse untermauerten außerdem die Anpassungsfähigkeit des Keims und könnten zumindest in Teilen erklären, warum dieser als Generalist auftrete, so Ponath. „Der opportunistische Erfolg von Fusobacterium nucleatum im Krebsgewebe ist wahrscheinlich darauf zurückzuführen, dass die Gene stets aktiv sind, die für die Adhäsion an die Tumorzellen verantwortlich sind“, sagt der Wissenschaftler.
Für die künftige Therapie von Krebserkrankungen ist die Kartografierung des Fuso-Transkriptoms eine wichtige Grundlage. In einem nächsten Schritt können Gene identifiziert werden, auf die eine Behandlung zielen, um das Fusobacterium in Karzinomen zu beseitigen und das Krebswachstum einzudämmen.
Originalpublikation:
Falk Ponath, Caroline Tawk, Yan Zhu, Lars Barquist, Franziska Faber, and Jörg Vogel: RNA landscape of the emerging cancer-associated microbe Fusobacterium nucleatum. Nature Microbiology, 08.07.2021. DOI: 10.1038/s41564-021-00927-7